< 서포트 벡터 머신 >
서포트 벡터 머신은 분류 과제에 사용할 수 있는 머신러닝 지도학습 모델
즉, 분류를 위한 기준 선을 정의하는 모델이며 분류되지 않은 새로운 점이 나타나면 어느 쪽에 속하는지 확인해서 분류 과제를 수행할 수 있게 됨
어떤 선이 데이터를 분류할 최적의 선인가?
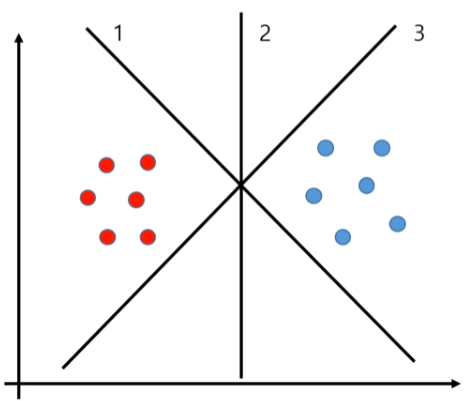
SVM은 결정 경계(경계선)을 어떻게 정의하고 계산하는지 이해하는게 중요함.
용어정리
- Support Vector : 경계선과 가장 가까이 있는 데이터 포인트
- 구분선(Decision Boundary) : 데이터를 구분하는 경계선
- 마진(Margin) : 구분하는 선과 Support Vector와의 거리
Support Vector(데이터 포인트) 마진을 최대화하여 구분선을 그으면, 예측하고자 하는 다른 데이터가 들어왔을 때
정확도가 더 높은 예측결과를 낼 수 있음
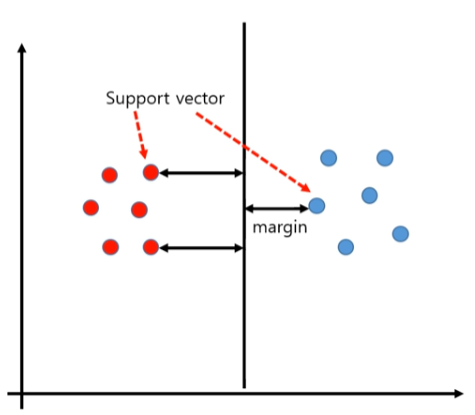
Margin을 최대화 하는것이 핵심!
- 강건도(Robustness) : 로버스트 한 것은 아웃라이어(Outlier)의 영향을 받지 않는 것, 튼튼한 정도
Ex. 1, 2, 3, 4, 5 중앙값 3, 평균 값 3 로버스트 하다
Ex. 1, 2, 3, 50, 105, 중앙값 3, 평균값 32.2 로버스트 하지 않다.
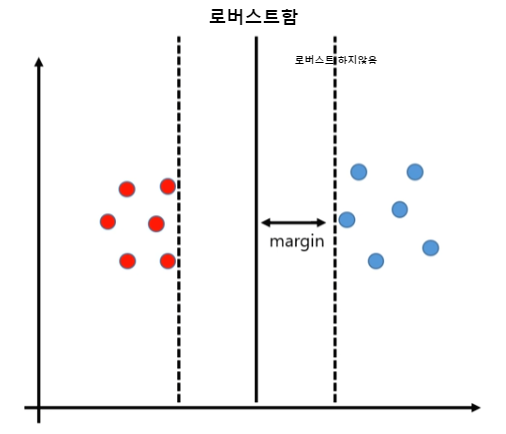
중간선이 제일 로버스트 해보임.
아웃라이어(Outlier) 처리
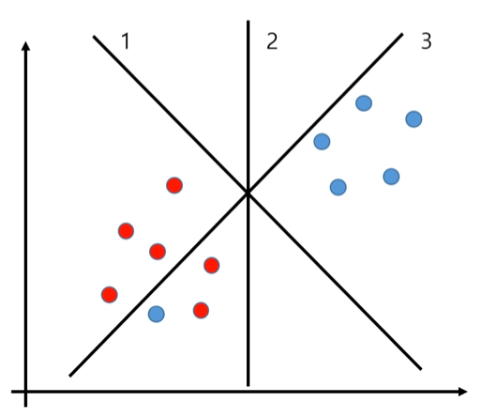
Outlier는 두 데이터 간의 구분을 지을때 최적의 선을 찾기위해 애매한 데이터들을 몇개 무시하고 선을 찾는 것
예를들면, 아래의 이미지에서 정확하게 구분할 수 있는 직선은 없다. 따라서, 빨간색 사이의 파란색 데이터를 outlier취급 해서 무시하고 Margin을 최대화하는 구분선을 찾는것
커널 트릭(Kernel Trick)
데이터를 구분하기 위해 직선을 그릴 수도 없고, Outlier처리를 할 수도 없는 상황 일 때.
아래의 이미지는 직선, Outlier처리 둘다 진행할 수가 없음.
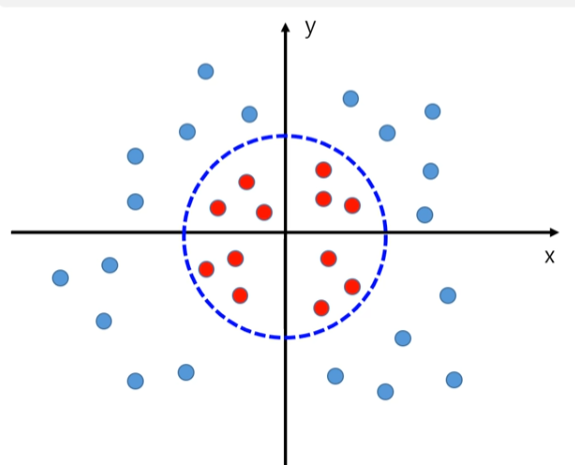
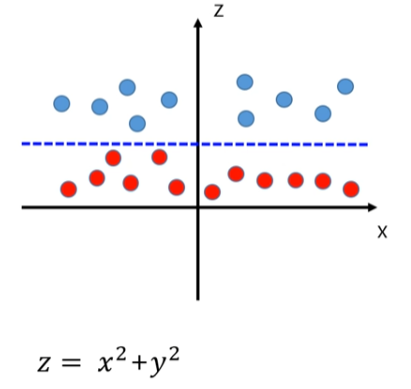
차원을 바꾸어 구분선을 그려야 한다
$ z = \sqrt{x^2 + y^2} $
z는 원점에서 부터 데이터의 점까지의 거리
이제 X의 특징과, Z의 특징을 이용해 차원을 바꿔 그래프를 그리면 구분선을 그을 수 있게된다.
< SVM을 이용한 당뇨병 구별>
1. 당뇨 데이터
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
diabdata = pd.read_csv("./data/diabetes.csv")
diabdata.head()2. 데이터 전처리
from sklearn.model_selection import train_test_split, GridSearchCV
Y_data = diabdata['Outcome']
X_data = diabdata.drop('Outcome', axis = 1)
X_train, X_test, Y_train, Y_test = train_test_split(X_data, Y_data, test_size=0.3)- Train과 Test를 7:3으로 분류
3. 모델학습
from sklearn.svm import SVC
svclassifier = SVC(kernel = 'linear')
svclassifier.fit(X_train, Y_train)
4. 검증
from sklearn.metrics import accuracy_score
from sklearn.metrics import classification_report, confusion_matrix
Y_pred = svclassifier.predict(X_test)
print("정확도 : ", accuracy_score(Y_test,Y_pred))
print(confusion_matrix(Y_test,Y_pred))
print(classification_report(Y_test,Y_pred))- 정확도 74% 정도
- Confusion_Matrix
[[126, 19]
[40, 46]]
4.1 피어슨 상관계수를 통한 분석
[머신러닝 수학] - 상관계수 정리 (피어슨 상관계수, 스피어만 상관계수 )
상관계수 상관계수? 산점도에서 점들이 얼마나 직선에 가까운지를 타나내는 척도를 상관계수 라고 한다. 아래 그림을 보며 설명을 하자면, p의 값이 $ X_1 X_2$ 특징에 대해서 얼마나 직선적인 경
yoon1seok.tistory.com
colormap = plt.cm.viridis
plt.figure(figsize=(15,15))
plt.title("Pearson Correlation of attributes", y=1.05,size=19)
sns.heatmap(diabdata.corr(),linewidths=0.1, vmax=1, square=True, annot=True)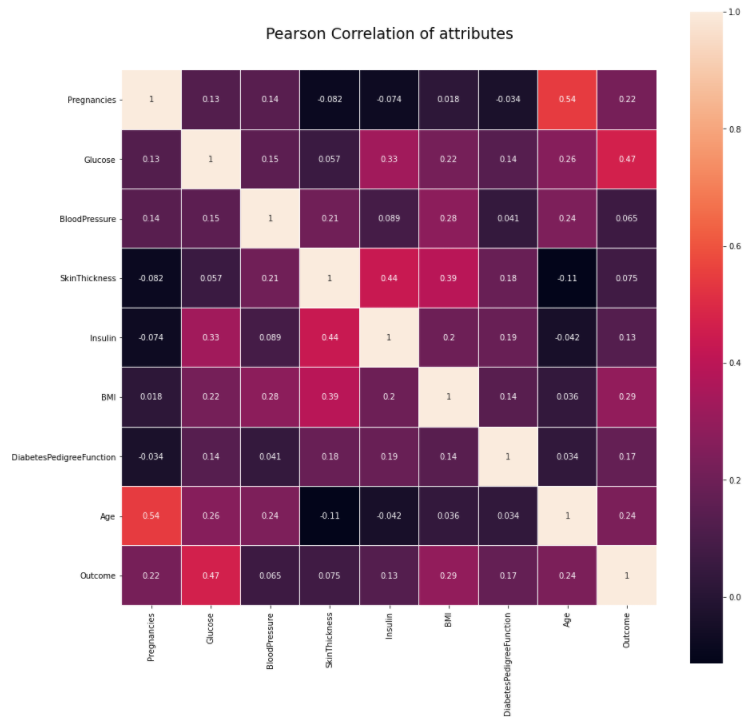
4.2 산점도 행렬
# KDE 커널밀도추정곡선
spd= pd.plotting.scatter_matrix(diabdata,c=diabdata['Outcome'], figsize=(20,15), diagonal="kde")
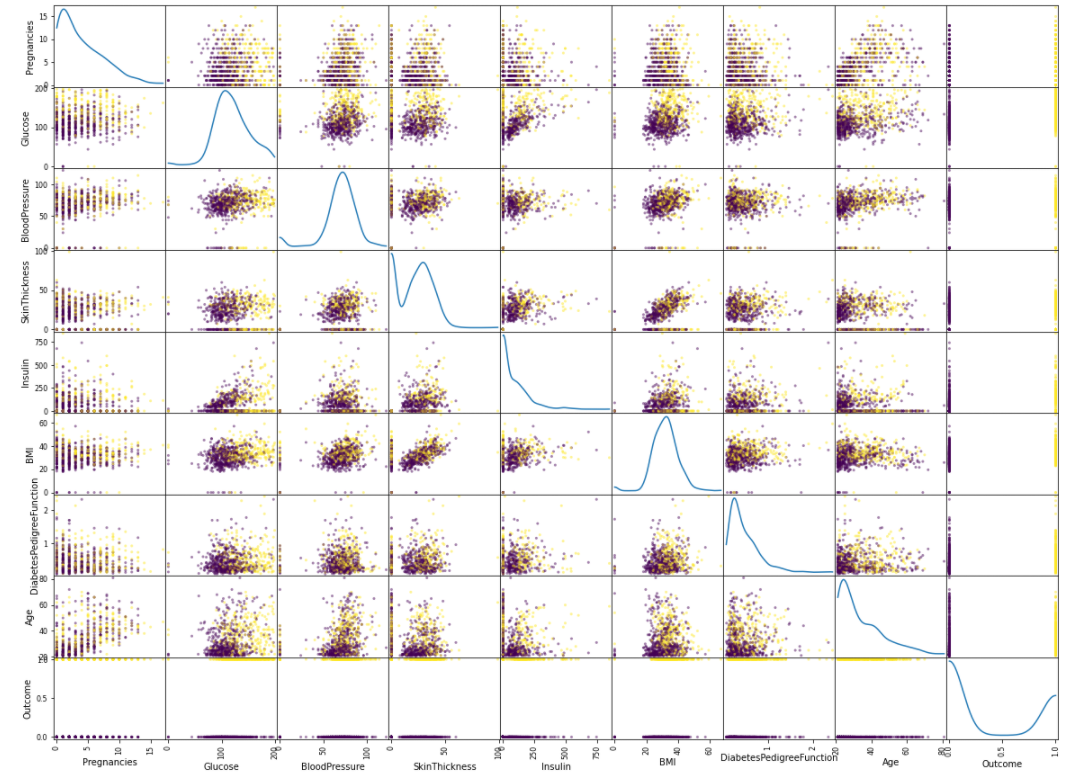
- 산점도 행렬을 이용해 'Outcome'을 기준으로 모든 특징들끼리 비교되는 것을 한번에 볼 수 있다.
- 산점도 행렬을 보면 노란색이 1은 당뇨를 가진 사람, 보라색이 0은 당뇨가 없는 사람인거 같은데 'Age' 행열을 보면 40~80대에 노란색이 밀집되어 있는 것을 볼 수 있고, 보라색이 비교적 0~40대에 많이 분포되어 있는것을 볼 수 있다.
속성중 diagonal = "kde"는 커널밀도추정곡선이라고 하는데 나도 뭔지 몰라서 정리하려고 한다..
삽질의 시작.. 몇 미터 까지 파려나
